Palindromati
fdocc arroba
yahoo punto com
Biotecnólogo Independiente.
RESUMEN
Este artículo describe a una familia de heterotrancritos
artificiales (quimeras de RNA), miles de secuencias en el Genbank que contienen fragmentos o al adaptador del tipo EcoRI ctcgtgccgaattcggcacgag actuando como enlazador palindrómico, el
cual une a dos o más genes producto de differentes cromosomas. Esto sucede
debido a que las metodologías actuales producen dichas secuencias que se
encuentran en el Genbank, así como en
los microarreglos de Affymetrix, y en
muchos artículos publicados que reportan o que usan dichas secuencias
portadoras del ligador del tipo EcoRI
dentro de sus regiones codficantes, y/o de sus sitios mRNA 5’UTR ó 3’UTRs. Este
ligando del tipo EcoRI y sus heterotranscritos
son considerados aquí como artefactos experimentales, caracterización que
pudiera ayudar a prevenir errores tanto en los estudios de mecanismos
moleculares así como en el descubrimiento de productos farmacéuticos.
Palabras Clave: EcoRI, Palíndroma, Perilipina, Genbank, Affymetrix, microarreglo (microarray),
especies-específico.
INTRODUCCIÓN
Es vital en el descubrimiento de nuevos tratamientos
médicos el enfocarse en moléculas específicas sin efectos colaterales en otros
tejidos. Para lograr dicho objetivo es necesario un estricto control de calidad
dentro de las bases de datos moleculares. Este artículo describe el hallazgo de
numerosos artefactos metodológicos reportados al Genbank. Se recomienda el más detallado análisis de secuencias de
ácidos nucleicos para nuevos descubrimientos biológicos, médicos y
farmacéuticos.
Un único RNA atando en su sola hebra a dos
diferentes genes procedentes de dos diferentes cromosomas humanos (1) fue el
principio teorético para mi estudio de heterotranscritos.
Aquí defino heterotranscritos
como quimeras, secuencias compuestas de fragmentos correspondientes a dos o más
genes procedentes del uno o de dos o más cromosomas.
Consideré que dicho fenómeno reportado en
referencia (1) debería de ser reflejado en una racional y lógica combinación de
productos genéticos Inteligentemente Diseñados (2, 3). Ya que la mayoría de las
moléculas y de las rutas biológicas vitales se encuentran presentes en muchos
organismos, inicialmente pensé que el fenómeno descrito en la referencia (1)
tal vez estuviera presente en muchas secuencias naturales como posible común
denominador funcional.
Inicialmente supuse que el estudio de secuencias
similares o relacionadas con la presentada en la referencia (1), quizás pudiera
ser útil en nuestro entendimiento de las bases moleculares de la variabilidad
biológica.
Así, éste fenómeno se perfilaba como posible
explicación para la abundancia de proteínas que excede el número de sus genes,
debido a múltiples combinaciones modulares entre sus diversos mRNAs. Recientes
estimados para humanos consideran que existen más de un millón de proteínas
producidas por solamente unos 20,000 ó 25,000 genes (4).
Con dichas consideraciones en mente, mi idea
inicial fue que, si la referencia (1) estaba en lo correcto, entonces la
producción de numerosas proteínas podría haber experimentado un putativo
proceso de hetero-ensamblaje de RNA al momento de su formación.
Sin embargo, después de cinco años de comparar
secuencias, he llegado a percibir que esas hetero-secuencias que usan a un
mismo oligonucleótido como su ligador común, son únicamente artefactos
metodológicos.
El elemento común de éstas quimaeras es el ligador (el "linker”)
ccgaattcgg (que se presentó en referencia 1 dentro
de la secuencia L21934 para la enzima ACAT-1
del H. sapiens). Esto hace que las
referencias 1, 5 y 6 (si describieran a un fenómeno real), se refieran a un
evento especies-específico único para
los seres humanos (2, 3).
Otro artículo con la misma secuencia (1) ha sido
publicado recientemente por ese mismo grupo (5). Sus autores mencionaron desde
la referencia (1) la similitud de ese ligador con el adaptador mas uado de EcoRI (herramienta extensamente usada en
investigación en biología molecular), así de que la puerta sigue abierta para
verificar si éste es un artefacto metodológico o no (5).
La construcción inicial de esa secuencia demostró que su
librería de cDNA fue transformada en E.
coli (cepa MC1061) usando el
vector fagémido pBluescript, así como el vector de expresión pcDNA. Después, de nuevo lo retransformaron en la misma cepa de E. coli (6). Sin embargo, recientemente he visto que el uso de vectores similares pudiera
estar involucrado en la producción de artefactos quiméricos en múltiples
instancias, como en los ejemplos presentados en los Cuadros 1 y 2 (7).
Una posible, aunque remota, explicación para la
referencia (1) es que allí estamos tratando con un proceso natural,
exclusivamente restringido a los seres humanos. Así de que, cualesquiera que
sea el veredicto final, el hecho es que el ligador o adaptor del tipo EcoRI descrito en (1) fue el punto
inicial para los siguientes hallazgos que se describen en éste artículo.
Mi hipótesis personal es que los heterotranscritos o
quimeras que incluyen al ligador palindrómico del tipo EcoRI ctcgtgccgaattcggcacgag o
sus secuencias relacionadas, que se extienden al menos por unas doce bases, son
artefactos artificiales debidos a las metodologías moleculares usadas, evento
mediado principalmente por la interacción vector-hospedador.
RESULTADOS Y
DISCUSIÓN
El hallazgo de
un palíndroma similar en los microarreglos (microarrays) de Affymetrix
Las bases para este artículo aparecieron mientras
trabajaba con los microarreglos. Estaba yo estudiando los cambios en la
expresión genética en los ratones resistentes a la obesidad con la perilipina
inactivada, "knock-out” (8, 11),
mediante el uso de los DNA-Chips de Affymetrix,
MG-U74A-v2 y usando el software gratuito educacional dChip V.1.2 (9). Entonces un particularmente intrigante
hetero-transcrito se presentó, la secuencia nucleotídica AB030505, inicialmente
reportada por sus remitentes como si fuera el mRNA del Mus musculus para UBE-1c1, UBE-1c2 y UBE-1c3 (cds completos). A
continuación se describe la secuencia AB030505 y su elemento común, el ligador
del tipo EcoRI, presente en miles de
secuencias del Genbank.
Un cuidadoso estudio de la secuencia de nucleótído
AB030505 usando Blast (10) me dirigió
hacia un elemento que estaba ligando a dos largas secciones de dos diferentes
genes en un RNA:
1. La secuencia de nucleótido AK078792
del cromosoma 10, codificando para un homólogo de la proteína mutada ubicua de
melanoma (Mum1) y
2. La secuencia de nucleótido BC036273
del cromosoma 12, que codifica para la retinol deshidrogenasa 11 (similar al
Arsdr1).
El elemento de enlace dentro de la secuencia AB030505
resultó ser el palíndroma ctcgtgccgaattcggcacgag, compuesto
por 22 nucleótidos.
Aquí también, así como en el reporte inicial (1), dos
transcritos originados en dos diferentes cromosomas aparecieron pegados en una
sola hebra de mRNA. 22 bases centrales conteniendo al ligador, el palíndroma ccgaattcgg,
similar al inicialmente reportado por la referencia (1).
Una secuencia palindrómica para la doble hélice de DNA
posee los mismos nucleótidos si se lee en la dirección de 5’ a 3’, que es la
dirección normal de lectura, ya sea desde la hebra plus (+) o la hebra minus
(-). Una evaluación manual y visual de éste ligador palindrómico se llevó a
cabo. Sorprendentemente, éste ligador se encontró presente en miles de
secuencias reportadas al Genbank.
El Cuadro 2 expandido (7) presenta numerosos ejemplos del
palíndromo (o secuencias relacionadas) que han sido reportados por sus
remitentes como si se encontrara dentro de las regiones codificantes. El
ligador palindrómico es frecuentemente traducido como el péptido artificial RAEFGT, ausente en las bases de datos
de secuencias de proteínas (10).
Incremento en el número de secuencias
palindrómicas reportadas al Genbank
Un
incremento mensual ha sido observado en el número de secuencias que contienen
al ligador del tipo EcoRI o sus
derivados dentro de miles de secuencias. En un ejemplo reciente (14 Oct. 2005)
efectuado en Blastn (alineamiento nucleótido - nucleótido), seleccionando
la base de datos para las secuencias de ácidos nucleicos no redundantes (nr) del Genbank, una consulta de 44 letras
palindrómicas se usó:
CTCGTGCCGAATTCGGCACGAGCTCGTGCCGAATTCGGCACGAG
Con esta secuencia obtuve 6010 Blast Hits usando las siguientes condiciones:
1. 106 fue el número mínimo esperado. Algunos de
esos resultados se presentan en el Cuadro 2 (7).
2. 1000 fue el número de descripciones y de
alineamientos.
En
la base de datos alterna del Genbank
conteniendo secuencias incompletas expresadas (expressed sequence tags, est),
que son mRNAs de posibles proteínas, el número de secuencias conteniendo al
adaptador palindrómico EcoRI se
cuenta también en los miles.
Palíndromas adicionales encontrados
mediante el uso de microarreglos
Secuencias
adicionales pertenecientes a éstos ligandos también se encontraron mediante el
estudio de los resultados de microarreglos disponibles en el Internet usando la
mencionada herramienta de software dChip
(9) junto a la base de datos de las secuencias usadas por Affymetrix. En e1 Cuadro 1 se dan ejemplos conteniendo a los
ligadores palindrómicos.
Affymetrix ha sido una exitosa metodología de
microarreglos, para evaluar por ejemplo la expresión genética en humanos, en
ratones y ratas. Sin embargo, tanto la presencia de los heterotranscritos
artificiales y/o de sus ligadores artificiales pudiera conducir a una representación errónea de su expresión
genética dentro de los tejidos, ya que el área bajo la curva se reduce
dramáticamente para esas secuencias genéticas.
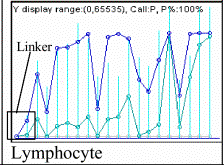
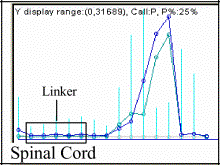
Cuadro 1. El ligador del tipo EcoRI se encuentra presente tanto en
secuencias del Genbank así como en
los microarreglos de Affymetrix para
humanos, ratones y ratas.
|
Clave/Organismo |
EcoRI Affymetrix Sondas del Genbank [DNA Chip] |
Gráfica con la no expresión del ligador tipo EcoRI |
Cita |
|
AB002533_at Homo sapiens |
Gaattcggcacgagcacgcgtgaga, Ggcacgagcacgcgtgagacttctc [en los DNA Chips Humanos HuGene-FL y Hu6800] |
Proteína Ribosomal LP2 (Qip1) |
Shipp et al, Nature Med. 8, 68. 2002 |
|
AJ243503 (99534_at) Mus musculus |
Ttcggcacgagctcgtgccggtcct [en los DNA Chips del Ratón MG-U74Av2 y MG-U74A] |
Adipocito Ghrelina |
Castro-Chavez et al. Diabetes 52, 2666. 2003 |
|
AI045710 (rc_AI045710_at) Rattus
norvegicus |
Atgatatgtacagatccctcgtgcc, tgatatgtacagatccctcgtgccg, tatgtacagatccctcgtgccgcct, tgtacagatccctcgtgccgcctcg, gtacagatccctcgtgccgcctcgt [en el DNA Chip de la Rata RG-U34B} |
Disúlfidoo isomerasa, prot. relacionada
(Erp70) |
Children's National Medical Center |
Nota: El palindróma ctcgtgccgaattcggcacgag
del tipo EcoRI causa la caída en la
expresión genética registrada por el microarray,
demostrando su ausencia en los organismos [dChip
V.1.2 (9)]. En la seguna columna se resaltan en azul claro las porciones
correspondientes al linker
palindrómico; en azul obscuro, los nucleótidos que son reemplazados para
obtener el segundo set de sondas en "mismatch"
para los DNA-Chips de Affymetrix.